Deux nouveaux projets COVID-19 labellisés à l’IDA

Ces projets, qui ont pour objectifs de développer de nouvelles méthodes pour inhiber l’entrée du virus dans les cellules (traitement), ou des nouvelles méthodes pour détecter ce virus (diagnostic), associent les laboratoires de biologie (LBPA) de chimie (PPSM) et de physique (LUMIN) de l’École, en collaboration avec des laboratoires de l’Université Paris-Saclay (LAMBE).
Projet 1 : la détection du virus
Ce projet propose de développer un dispositif pour la détection du virus et ainsi améliorer les tests COVID-19, en mettant en œuvre une amplification isotherme de l’ARN viral sur un dispositif microfluidique en gouttes (microfluidique digitale) ». Les chercheurs espèrent ainsi détecter des faibles charges virales, qui seront détectées optiquement dans des microgouttes en flux (à très haut débit) et électriquement à l’aide d’un nanopore (orifice de quelques dizaines de nanomètres, ayant la taille d’un virus).
Les objectifs
Les tests du COVID-19 fonctionnent sur l’analyse d’un échantillon prélevé chez le patient afin de voir si des ARN correspondent au génome du virus.
Ce nouveau projet a pour but d’améliorer les tests COVID-19 en utilisation une nouvelle méthode d’amplification appelée « RPA en microfluidique en goutte (digitale) » capable de détecter des faibles charges virales.
La possibilité de contrôler la qualité de l’amplification d’une séquence virale par "PCR en gouttes" (dPCR) permettrait également de dépasser les limitations des tests COVID classique en se basant sur la technologie « microfluidique en gouttes » pour la compartimentation de molécules d’ADN ou d’ARN.
Cette nouvelle approche permettrait de produire des tests plus rapides et peu chers qui pourraient être utilisés à grande échelle, et en particulier dans les pays en voie de développement, de pathogènes tel que le corona virus.
En quoi consiste la méthode d’Amplification RPA (Recombinase Polymerase Amplification) digitale et les techniques de microfluidique ?
Il s’agit d’une technique récente d’amplification de l’ADN ou de l’ARN qui, contrairement à la technique d’amplification PCR standard, s’opère à température constante (autour de 40 °C). Elle permet ainsi une amplification simple et rapide de séquences cibles d’ADN ou d’ARN dans des systèmes microfluidiques.
Les équipes
Le Laboratoire Lumière Matière et Interfaces (IDA-LUMIN)
L’équipe « Microsystèmes fluidiques et électriques pour l'étude du vivant » du laboratoire LUMIN est spécialiste de la conception de microsystèmes fluidiques pour les applications biologiques et médicales.
- Abdel EL ABED (coordinateur du projet)
- Bruno LE PIOUFLE (Institut d’Alembert – ENS Paris-Saclay)
L'équipe "Biophotonique et interactions moléculaires" , IDA-LBPA possède une expertise reconnue dans le domaine de la dynamique des biomolécules par des approches de fluorescence résolues en temps. Elle apportera son son savoir-faire sur la culture de cellules infectées et la production virale, son expertise sur les virus à ARN et les méthodes de quantification du génome viral.
- Éric DEPREZ (CNRS)
- Olivier DELELIS (CNRS)
- Clémence RICHETTA (ENS Paris-Saclay)
- Frédéric SUBRA (IR CNRS)
Le LAMBE , laboratoire CNRS de l’Université Paris Saclay situé à l’Université d’Evry apporte son expertise dans la détection électrique à l’échelle de la molécule unique du produit de la PCR par nanopore et dans l’analyse du signal.
- Laurent BACRI
- Nathalie JARROUX
- Juan PELTA
- Manuela PASTORIZA-GALLEGO
- Benjami CRESSIOT
Projet 2 : la recherche de molécules pour inhiber l’entrée du virus dans les cellules
La pandémie virale actuelle liée à la maladie COVID (COronaVIrus Disease) est causée par le virus SARS-CoV-2, un nouvel agent viral découvert au début de l’année 2020 en Chine.
Il fait partie de la famille des coronavirus (sous famille des betacoronavirus), une famille de virus à ARN qui cause des rhumes et des gastroentérites de nature bénigne chez l’homme et de nombreux animaux. Néanmoins, depuis le début du 21eme siècle, plusieurs coronavirus (SARS-CoV-1, MERS), provoquant des maladies respiratoires mortelles chez l’homme, sont apparus dans le cadre d’événement appelés zoonoses (passage de souches virales de l’animal à l’homme).
La recherche actuelle sur le SARS-CoV-2 peut donc s’appuyer sur les données et les hypothèses déjà émises pour d’autres coronavirus et notamment le SARS-CoV-1 qui est son cousin le plus proche parmi les virus humains et qui est apparu en Chine en 2002.
Il avait ainsi été découvert en 2006 que la “porte d’entrée” du virus dans les cellules humaines (et notamment dans les cellules alvéolaires des poumons et les cellules endothéliales cardiaques) pour le SARS-CoV-1 était l’enzyme de conversion de l’angiotensine II (appelé ACE2), il a été récemment montré qu’il en était de même pour SARS-CoV-2.
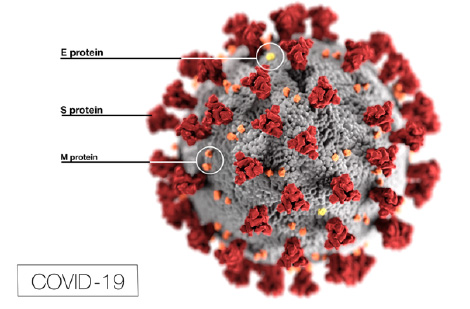
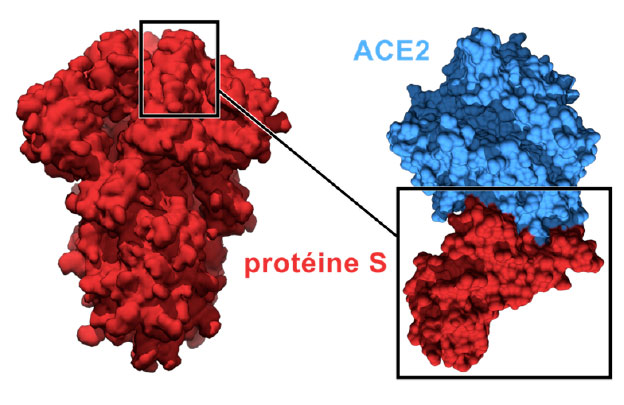
La reconnaissance se fait par l’intermédiaire de la protéine S (protéine de spicule ou Spike protein, en rouge dans la figure) une grosse protéine située à la surface de la particule du virus, cette protéine est commune à tous les coronavirus et leur donne leur aspect “couronné” d’où leur nom.
L’entrée du virus dans les cellules humaines repose de façon essentielle sur l’interaction entre la protéine S et le récepteur ACE2. Cette interaction est très forte et sa mesure nécessite des appareillages très particuliers dont nous disposons au laboratoire, il s’agit des appareils de type SPR (Surface Plasmon Resonance) qui utilisent des méthodes de détection optiques capables d’identifier clairement les phases d’association et de dissociation des complexes.
De façon intéressante, il a été montré que l’affinité du nouveau virus pour ACE2 a augmenté de façon significative par rapport au SARS-CoV-1, et il a été suggéré que le pouvoir infectieux supérieur du SARS-CoV-2 par rapport au SARS-CoV-1 pourrait être liée à cette affinité accrue.
L’analyse de ces interactions et leurs quantifications est importante pour mieux comprendre les composantes les plus importantes de l’interaction au niveau moléculaire, et surtout identifier des molécules capables de perturber ces interactions.
Comme pour beaucoup de virus, l’entrée du SARS-CoV-2 dans les cellules cibles est déterminée par des contacts spécifiques entre des protéines situées à la surface du virus (protéine S) et certaines protéines localisées sur les faces externes des cellules.
Pour le SARS-Cov-2, la protéine positionnée sur la face externe des cellules qui est utilisée est l’enzyme de conversion de l’angiotensine II appelée ACE2. Cette enzyme est par ailleurs très connue pour son rôle dans le contrôle de l’angiotensine, un peptide impliqué dans la régulation de la pression dans les vaisseaux sanguins. Précisons qu’il existe deux enzymes de conversions de l’angiotensine, ACE1 (ciblé par les médicaments hypertenseurs) et ACE2.
Si l’entrée du virus dans les cellules se fait par l’interaction ACE2-protéine S, le blocage de cette interaction ou son empêchement par des ligands organiques ou peptidiques peut constituer une voie d’inhibition du virus.
L'objectif du projet
Ce projet a pour objectif de mettre au point des méthodologies permettant l’identification de telles molécules via une plateforme de recherche de ligands interférant avec l’interaction entre la protéine de Spicule (Protéine S) du SARS-CoV-2 et l’enzyme de conversion à l’angiotensine ACE2).
Il s'agit de caractériser en détails les interactions entre ACE2 et la protéine S, d’étudier l’impact de mutations des domaines de la protéine S et de tester de nouvelles molécules pour leur capacité à perturber cette interaction afin d’empêcher l’entrée du virus dans nos cellules.
Le projet doit se dérouler sur deux ans. L'aide de l’Institut d'Alembert (IDA) est une opportunité pour permettre d’obtenir des résultats et confirmer l’intérêt de l’utilisation d’une plateforme SPR dans la détection de molécules prometteuses.
Les équipes
Il est porté par Olivier MAUFFRET (IDA-LBPA) et Joanne XIE (IDA-PPSM).
Le laboratoire LBPA va, d’une part, utiliser ses compétences en biologie moléculaire et en biophysique (SPRi, RMN) pour produire les protéines partenaires de l’interaction et établir les protocoles permettant des descriptions fiables de leurs interactions. D’autre part, des approches de biologie computationnelle seront mis au point pour guider la conception rationnelle d’inhibiteurs de l’interaction sur la base de la structure tridimensionnelle des protéines et des résultats des expériences.
- Claude NOGUES,
- Brahim HEDDI,
- Loussiné ZARGARIAN,
- Olivier MAUFFRET,
- Marco PASI
Le PPSM va utiliser ses compétences en synthèse organique et peptidiques pour concevoir et synthétiser de nouveaux ligands sur la base de certaines molécules dont l’efficacité sur les interactions ACE2-protéine S du coronavirus de 2002, le SARS-CoV-1 a été montrée.
- Joanne XIE,
- Stéphane MAISONNEUVE